14 Sep Une nouvelle approche pour adapter la thérapie contre le cancer : exploiter les signaux des cellules cancéreuses
Adapter les médicaments en fonction des tumeurs pourrait mener à un traitement personnalisé et à de nouvelles thérapies.
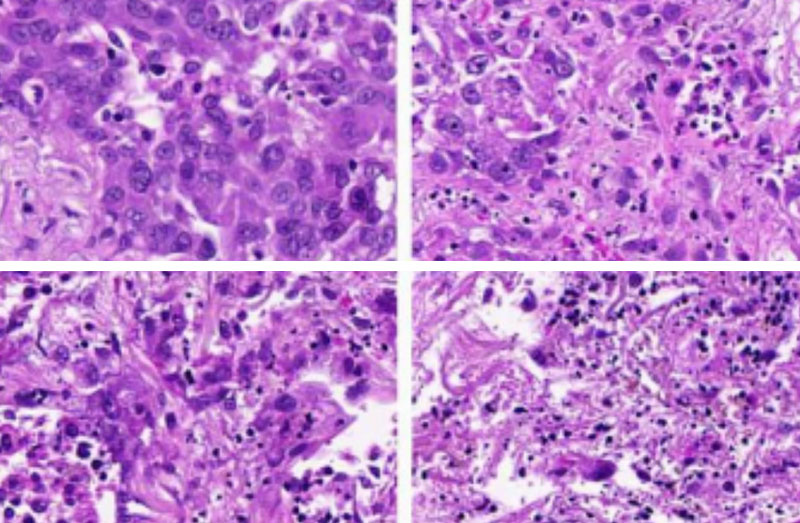
Échantillons de cancer du poumon (de gauche à droite) : cancer non traité (première image) ; les tissus des poumons ne répondent pas au médicament inhibant les microtubules (deuxième image) ; ni au médicament favorisant l’apoptose (troisième image) ; mais quand les deux médicaments sont utilisés en même temps sur le même tissu, les cellules cancéreuses meurent (quatrième image)
Choisir le bon traitement pour chaque patient atteint d’un cancer est la clef de son efficacité mais les médecins n’ont actuellement que peu d’indications leur permettant de concevoir le protocole de traitement ad hoc. Des chercheurs de l’Institut Weizmann des Sciences et du Broad Institute du MIT et d’Harvard ont développé une nouvelle méthode permettant de sélectionner la meilleure thérapie médicamenteuse pour traiter une tumeur donnée en se basant sur un score attribué aux signaux internes des cellules. En plus d’aider les médecins à faire le bon choix parmi les traitements existants, cette méthode peut aider à identifier de nouvelles cibles moléculaires pour développer de nouveaux médicaments. Ainsi, les chercheurs ont déjà utilisé cette approche d’identifier un gène cible pour traiter efficacement des cancers du sein avec mutation du BRCA. Cette étude a récemment été publiée dans Nature Communications.
La méthode la plus classique pour faire correspondre un médicament à une tumeur est de cibler une mutation précise dans les cellules tumorales. Malheureusement, la présence de telles mutations ne garantit pas l’efficacité du médicament, et en tout état de cause, de nombreux médicaments ne tiennent pas compte des mutations. Des chercheurs ont également tenté de prédire l’efficacité d’un médicament en analysant l’expression de certains gènes dans une tumeur mais très peu de gènes se sont avérés utiles dans le choix d’un traitement.
Au cours d’une nouvelle étude, deux laboratoires – l’un dirigé par le docteur Ravid Straussman du département de biologie cellulaire et moléculaire de l’Institut Weizmann et l’autre par le professeur Gad Getz du Broad Institute de Cambridge, Massachusetts – se sont associés pour créer une approche plus efficace utilisant les bases de données importantes sur le cancer rendues accessibles ces dernières années. Cette nouvelle approche ne se fonde pas sur les mutations ou sur des gènes particuliers mais sur les voies de signalisation : les chaînes de signaux biochimiques qui transmettent des messages cellulaires cruciaux, comme par exemple si une cellule doit se diviser ou croître ou si son métabolisme doit être modifié. Des méthodes avancées sont nécessaires pour analyser l’activité de ces chaînes car de nombreux gènes s’expriment dans les cellules dans le but de transmettre ces messages par toutes les voies possibles.
Le docteur Rotem Ben-Hamo, en postdoctorat, a analysé de grandes bases de données internationales contenant des informations sur l’expression de tous les gènes de près de 460 lignées de cellules cancéreuses provenant de dix types de cancers différents. En utilisant PathOlogist, un outil bio-informatique avancé développé par le professeur Sol Efroni de l’Université Bar-Ilan, les chercheurs ont attribué à chaque n voie de signalisation une note d’activité qui prend en compte le niveau d’expression des gènes, les connaissances que nous avons sur la structure de ces voies, les interactions qu’ont les gènes sur ces voies et si un gène donné bloque ou favorise la voie du message. Les scientifiques ont ensuite corrélé ces scores avec les données contenant des informations sur la sensibilité de différentes cellules cancéreuses à environ 500 médicaments anti-cancéreux différents.
Ils ont découvert que les notes d’activité de certaines voies leur permettaient de prédire si un cancer en particulier serait sensible à un médicament précis. En d’autres termes, les chercheurs ont créé un profil de tissus cancéreux indiquant aux médecins les meilleurs médicaments pour éradiquer les tumeurs. Ils ont même réussi à faire de telles prédictions pour près de 30 médicaments existants. Par exemple, quand des cellules de certains cancers des poumons avaient une note élevée pour une voie déclenchant l’apoptose – une forme de suicide cellulaire – elles étaient sensibles à une catégorie de médicaments connus pour inhiber les microtubules.
Puis, les scientifiques ont montré qu’ils pouvaient utiliser leurs connaissances sur ces voies de signalisation pour modifier la réponse des cellules à un médicament. Ils ont commencé par échantillonner des tissus de poumons chez un patient atteint d’un cancer ; d’après leur analyse, les voies favorisant l’apoptose n’étaient pas particulièrement actives dans ces tissus. Cela a été confirmé par leurs tests : ces tissus étaient résistants aux médicaments inhibant les microtubules. Mais quand les chercheurs ont ajouté, en plus d’inhibiteurs de microtubules, une substance augmentant l’activité de la voie de l’apoptose, les cellules cancéreuses ont été tuées.
Lors d’analyses plus poussées de ces données, les chercheurs ont corrélé les notes d’activité des voies de signalisation avec un autre type d’information : l’identité des gènes jouant un rôle si essentiel dans de nombreuses tumeurs que les bloquer ou les mettre sous silence peut tuer la tumeur. Ils ont découvert que les notes d’activité des voies de signalisation les aidaient ici aussi à identifier ces gènes « sensibles » dans un grand nombre de tumeurs. Par exemple, ils ont découvert que les tumeurs du sein avec une activité particulière dans la voie du BRCA – qui est corrélé avec la présence d’une mutation du BRCA – étaient extrêmement dépendantes de l’activité d’un gène appelé MAD2L1. L’analyse bio-informatique prédisait que mettre sous silence ce gène pourrait entraîner la mort des cellules tumorales chez les patients présentant une mutation du BRCA. Cette prédiction peut servir de point de départ pour la recherche de médicaments nouveaux ou existants permettant de traiter ce cancer du sein dévastateur.
Ainsi, les découvertes de cette étude suggèrent que les voies de signalisation pourraient servir de marqueurs biologiques prédictifs pour la médecine personnalisée du futur, aidant les médecins à savoir quel patient répondra à quel médicament. De plus, les voies de signalisation pourraient aider les chercheurs à identifier les talons d’Achille de nombreuses tumeurs orientant ainsi le développement de nouveaux médicaments.
Différents chercheurs ont participé à cette étude : le docteur Adi Jacob Berger, le docteur Nancy Gavert et le docteur Yaara Zwang du laboratoire du docteur Straussman dans le département de biologie cellulaire et moléculaire de l’Institut Weizmann ; le docteur Mendy Miller du laboratoire du professeur Getz du Broad Institute de Cambridge, Massachusetts ; le docteur Guy Pines du Centre médical Kaplan ; le docteur Roni Oren du département des ressources vétérinaires de l’Institut Weizmann ; le professeur Eli Pikarsky et le docteur Tzahi Neuman de l’Université hébraïque de Jérusalem ; et le professeur Cyril H. Benes de l’Hôpital général du Massachusetts.
Les recherches du docteur Ravid Straussman sont financées par le Centre intégré Moross pour le Cancer ; le Centre pour la microbiologie de la famille Knell ; le fonds de dotation pour la biologie de Maurice et Vivienne Wohl ; le fonds de recherche pour l’humanité des familles Fabrikant-Morse ; le fonds de recherche du docteur Chantal d’Adesky Scheinberg ; la fondation Rising Tide ; et le Conseil européen pour la recherche. Le docteur Straussman est détenteur de la chaire Roel C. Buck pour le développement de carrière.


