
08 Nov La chasse aux virus géants qui s’attaquent aux algues minuscules
Une menace mystérieuse rôde dans les océans, menaçant la prolifération des algues. Les chercheurs de l’Institut Weizmann ont mis au point un nouveau moyen de traquer le coupable – les virus géants – et d’identifier leurs traces dans des types spécifiques d’algues minuscules qu’ils attaquent.
On disait qu’ils venaient de l’espace et on affirmait même qu’ils étaient en fait des bactéries et qu’ils remettaient en cause la définition même des virus. Les virus géants, surnommés « girus », contiennent d’énormes quantités de matériel génétique – jusqu’à 100 fois plus que les autres virus – et certains sont plus grands que certaines bactéries. Ils étaient pratiquement inconnus de la science jusqu’au début du 21e siècle, mais les découvertes faites au cours des deux dernières décennies suggèrent qu’ils peuvent avoir un impact majeur sur la vie sur Terre.

(g-d) Dr. Daniella Schatz, Prof. Assaf Vardi and Amir Fromm
Les virus géants qui peuplent les océans infectent, entre autres, diverses espèces d’algues unicellulaires, des organismes photosynthétiques responsables d’environ la moitié de la production d’oxygène de la Terre et d’environ la moitié de la fixation du carbone à l’échelle mondiale. L’infection virale peut provoquer un effondrement rapide des efflorescences algales – des accumulations d’algues qui s’étendent sur des dizaines de milliers de kilomètres dans l’océan – ce qui peut, à son tour, affecter considérablement de vastes écosystèmes marins, atmosphériques et terrestres. Cependant, nous en savons encore très peu sur les hôtes naturels des virus géants, c’est-à-dire sur les espèces d’algues qui sont infectées par chaque type de virus.
Dans une nouvelle étude, des chercheurs du laboratoire du professeur Assaf Vardi, du Département des Sciences Végétales et Environnementales de l’Institut Weizmann des Sciences, ont utilisé le séquençage de l’ARN de cellules uniques pour analyser des échantillons prélevés dans une efflorescence algale dans les fjords de Norvège. Cela leur a permis de cartographier, avec un niveau de détail sans précédent, les relations entre les virus géants et les organismes marins unicellulaires, y compris les algues, que chacun d’entre eux infecte.
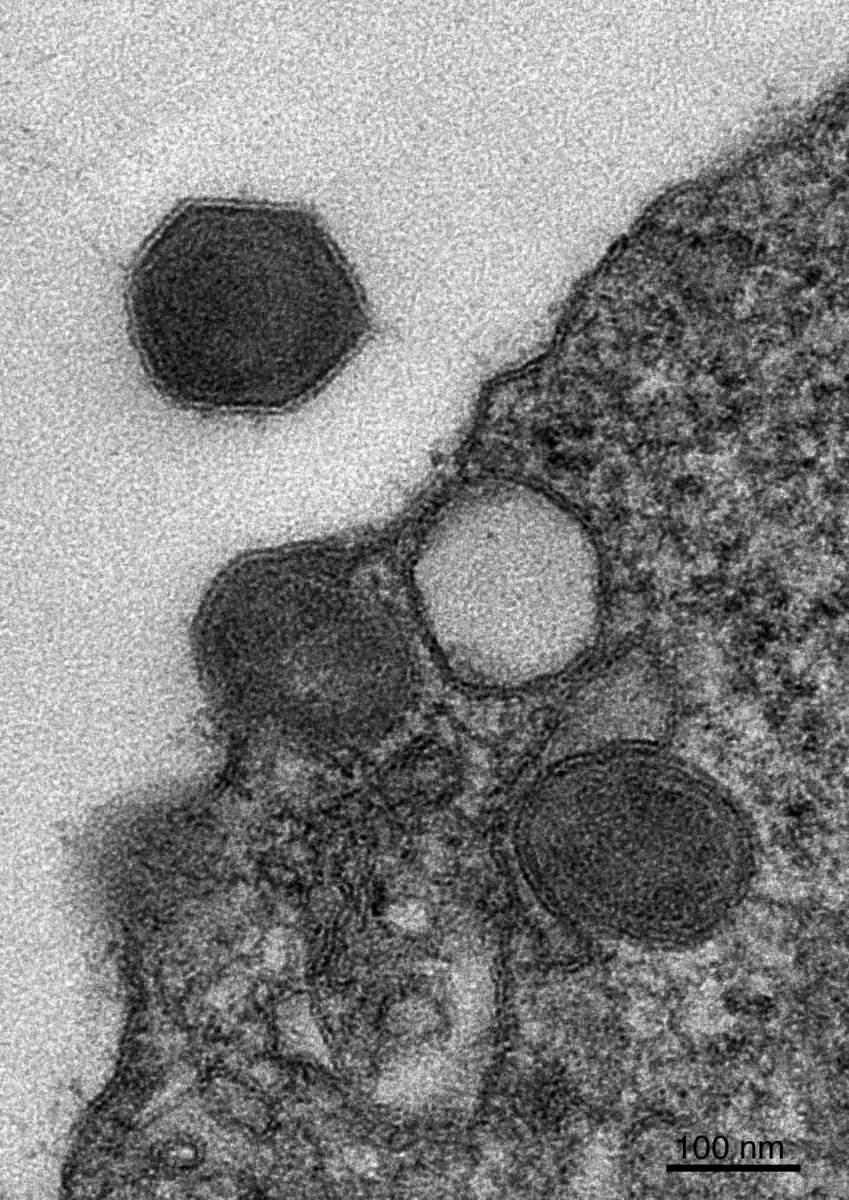
Comment les efflorescences s’effondrent : Quatre virus géants en forme d’hexagone qui se sont multipliés dans une algue unicellulaire (texture rugueuse à droite) sont sur le point d’infecter d’autres cellules d’algues. Trois virus sont intacts et prêts à agir, tandis que l’un d’entre eux (en blanc) est dépourvu d’ADN. Photo : Dr : Daniella Schatz
Jusqu’à récemment, le moyen le plus efficace d’analyser les populations de virus et d’algues dans l’océan consistait à les examiner en vrac, c’est-à-dire à analyser tout le matériel génétique isolé à partir d’échantillons d’eau de mer grouillant de virus et d’organismes marins unicellulaires. « Ces études ont fait progresser nos connaissances sur la répartition des algues et des virus dans l’eau, mais il manquait encore beaucoup d’informations », explique le Professeur Vardi. « Nous avons pu savoir quelles espèces étaient abondantes dans un échantillon donné, mais nous n’avons pas pu identifier les infections actives, ce qui a limité notre capacité à mesurer l’impact des virus sur la prolifération ou l’effondrement des algues. De même, nous n’avons pas pu identifier les populations moins répandues dans l’échantillon, ni déterminer l’hôte naturel de chaque virus, ni savoir si les virus étaient actifs dans les cellules. »
Pour surmonter ces limites et examiner les interactions qui existent dans l’environnement naturel, les chercheurs se sont rendus dans les fjords norvégiens, où ils ont provoqué une prolifération d’algues en ajoutant des nutriments à l’eau et en reproduisant les conditions qui conduisent à la prolifération. De retour au laboratoire, ils ont séquencé, cellule par cellule, l’ARN présent dans les cellules de leurs échantillons. C’est ce que l’on nomme « séquençage d’ARN de cellules uniques ». Les molécules d’ARN ainsi identifiées indiquent quels gènes sont actifs dans chacune des cellules des organismes échantillonnés à un moment donné. Cette méthode avancée et à haute résolution diffère du séquençage normal de l’ARN, qui consiste à rassembler l’ARN de tous les organismes de l’échantillon. Elle permet de marquer des cellules individuelles et d’identifier l’ARN qui apparaît uniquement dans cette cellule particulière.

Faire fleurir les fjords : L’équipe de recherche du laboratoire du professeur Assaf Vardi prélève des échantillons en Norvège.
Grâce à cette méthode, les chercheurs ont pu identifier les gènes actifs dans les cellules et, en les comparant aux bases de données existantes, déterminer à quelle espèce appartenait chaque cellule de l’échantillon. Mais ils ne se sont pas contentés de cartographier les hôtes ; ils ont également voulu déterminer quels virus infectent chaque cellule dans l’environnement naturel. Amir Fromm, l’étudiant en doctorat du laboratoire du professeur Vardi qui a dirigé l’étude, explique : « Nous avons réalisé qu’en analysant méticuleusement l’ARN, nous pouvions produire deux types de données, non seulement en identifiant les espèces d’algues, mais aussi en déterminant si les algues avaient été infectées par un virus géant et, le cas échéant, lequel. »
Avant cette étude, il n’existait aucune base de données classant les virus géants qui infectent les algues. Les chercheurs ont donc contacté le professeur Frank Aylward, expert en évolution des virus à Virginia Tech, qui, avec sa propre équipe, a constitué une base de données unique des virus géants, sur la base de l’ARN extrait des cellules.
Sur les dizaines de milliers de cellules examinées, beaucoup appartenaient à l’algue dominante de cette efflorescence, Emiliania huxleyi. Dans une étude antérieure, Vardi et son équipe avaient déjà identifié le virus géant spécifique qui infecte cette algue. Cette fois, ils se sont concentrés sur d’autres couples virus-hôte, présents en quantités infimes dans les échantillons. Au total, ils ont trouvé 972 cellules infectées, dont 71 n’appartenaient pas à l’algue dominante de l’efflorescence et qui comprenaient plusieurs combinaisons virus géant-hôte inconnues jusqu’à présent.
Ils ont ainsi découvert que le virus Imitervirales-07 infecte les cellules d’algues de la famille des Katablepharidaceae, un lien qui n’avait jamais été décrit auparavant. En examinant l’ARN du virus dans ces cellules, ils ont constaté qu’il produit des protéines qui ont un impact direct sur le destin des cellules, y compris des protéines capables de provoquer l’apoptose, ou suicide des cellules (mort cellulaire programmée) pendant la phase tardive de l’infection. Les chercheurs ont même trouvé une arme fatale : Ils ont mis en évidence un lien entre l’apparition du virus dans les échantillons examinés et l’effondrement de la population d’algues hôtes peu de temps après.
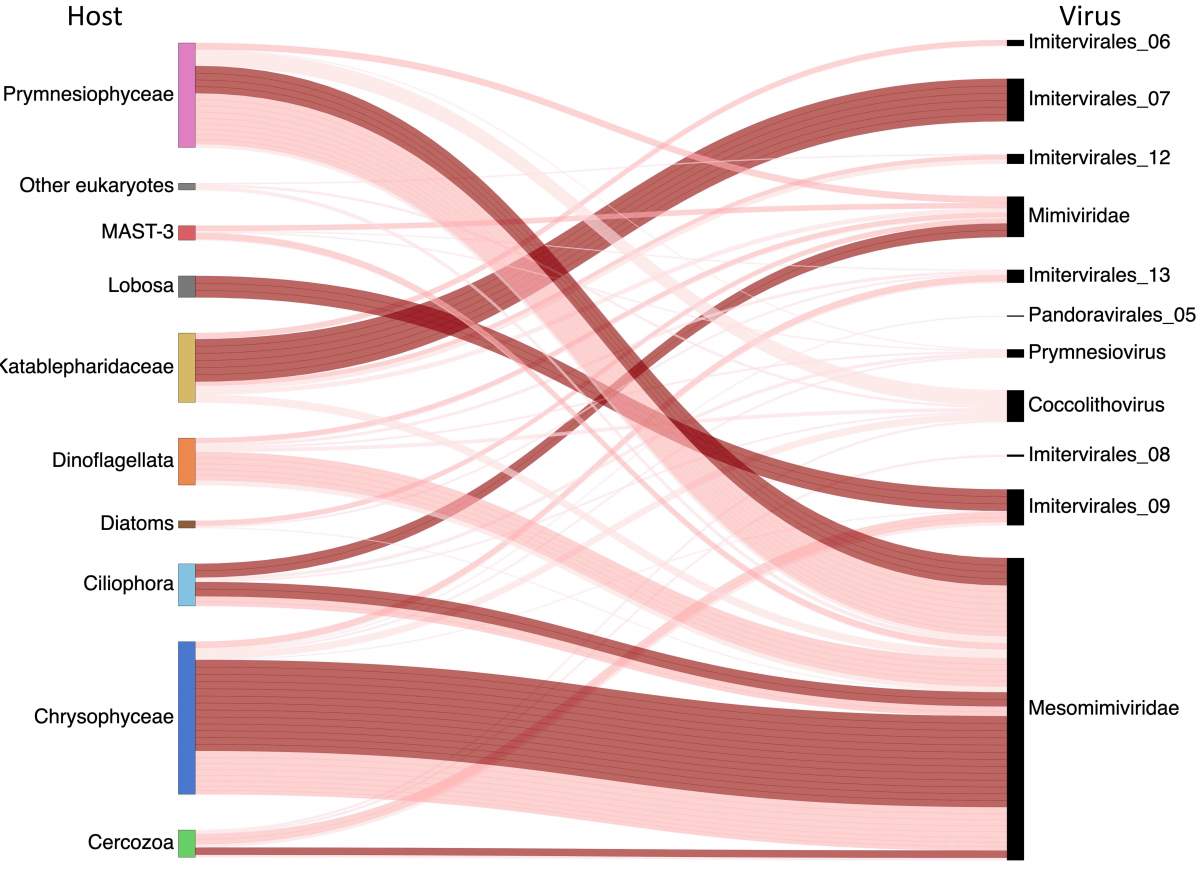
La ligne qui relie un virus géant à une algue minuscule : familles d’algues unicellulaires (à gauche) et virus géants (à droite) qu’elles hébergent, tels que découverts dans l’étude.
Comme les chercheurs ont réussi à relever le défi informatique de la caractérisation des cellules infectées dans des échantillons à partir de quantités infimes d’ARN, ils pensent que leur méthode peut également être utilisée pour identifier les interactions des algues avec des virus et d’autres agents pathogènes dans d’autres parties du monde, y compris dans des environnements climatiques extrêmes tels que les pôles et les lacs alpins. « En raison des processus liés au changement climatique, d’anciens agents pathogènes sont libérés par la fonte des calottes glaciaires aux pôles. Cette nouvelle méthode nous permettra d’identifier ces agents pathogènes beaucoup plus facilement et d’aider l’humanité à se préparer à la menace environnementale qu’ils peuvent représenter », explique M. Vardi.
La Science en Chiffres
Les scientifiques pensent qu’il y a 1031 virus rien que dans les océans, soit plus de 10 milliards de fois le nombre estimé d’étoiles dans l’univers visible.


