
17 Nov Les biopsies liquides : le diagnostic du cancer à partir d’un millilitre de sang
Si des études plus importantes confirment les résultats d’une innovation de l’Institut Weizmann, le diagnostic du cancer pourrait un jour être aussi simple qu’une prise de sang…
Les tests sanguins – simples, non invasifs et économiquement réalisables – promettent de devenir la prochaine étape majeure du diagnostic du cancer. Cependant, la plupart de ces tests, appelés biopsies liquides, ne sont actuellement pas assez fiables pour être utilisés à grande échelle. Une nouvelle approche multiparamétrique mise au point à l’Institut Weizmann des Sciences pourrait déboucher sur un test sanguin permettant de diagnostiquer le cancer avec une précision sans précédent. Cette recherche est publiée dans Nature Biotechnology.
« La plupart des méthodes conventionnelles disponibles aujourd’hui en clinique pour détecter et diagnostiquer le cancer sont invasives et désagréables », explique le Dr Efrat Shema du Département d’Immunologie et de Biologie Régénérative de Weizmann, qui a dirigé l’équipe de recherche. Le prélèvement d’échantillons par biopsie au moyen d’une aiguille, d’une endoscopie ou d’une intervention chirurgicale peut être douloureux et parfois risqué, et les méthodes d’imagerie, telles que l’IRM ou la tomographie par émission de positons, nécessitent des équipements coûteux et encombrants qui ne sont pas disponibles partout. Des tests sanguins efficaces pour le dépistage ou le diagnostic du cancer pourraient constituer une alternative intéressante. « En éliminant l’inconfort, les gens seraient moins enclins à éviter de se faire dépister – et plus susceptibles de voir leur cancer détecté plus tôt », explique Vadim Fedyuk, qui a dirigé l’étude avec son collègue Nir Erez, étudiant diplômé.

(de gauche à droite) Nir Erez, Dr. Noa Furth, Dr. Efrat Shema and Vadim Fedyuk
L’idée de diagnostiquer le cancer à l’aide de biopsies liquides est née du fait que le sang contient de l’ADN et des protéines flottant librement, excrétés par les cellules sanguines mortes chez les personnes en bonne santé – et chez les patients cancéreux, par les cellules tumorales mortes également. « Certains des sous-produits de la destruction des cellules, y compris l’ADN et les protéines du cancer, sont déversés dans la circulation sanguine, et nous savons comment les collecter et les analyser », explique le docteur Shema.
Un certain nombre de tests sanguins pour le cancer sont déjà à un stade avancé de développement, mais la plupart présentent des inconvénients qui peuvent limiter leur utilisation. Lorsque les premiers tests de ce type ont été mis au point, ils recherchaient des signes génétiques de cancer, c’est-à-dire des mutations, mais celles-ci peuvent être difficiles à repérer car les segments mutés ne représentent qu’une petite fraction de l’ADN en circulation. De plus, ces mutations ne conduisent pas toujours à un cancer et peuvent également être présentes chez des personnes en bonne santé. Plus récemment, les méthodes de biopsie liquide ont commencé à s’appuyer sur l’épigénétique, c’est-à-dire sur des modifications du génome de la cellule qui n’impliquent pas de mutations de l’ADN – par exemple, des étiquettes chimiques qui se fixent à la molécule d’ADN et modifient l’expression des gènes. Ces approches ont également rencontré des difficultés, soit parce qu’elles nécessitent des quantités excessives de sang, soit parce qu’elles recherchent un seul changement épigénétique qui ne peut donner des résultats suffisamment fiables.
Dans la nouvelle étude, le docteur Shema a entrepris de repenser cette analyse épigénétique, en cherchant à en développer une qui s’appuierait sur un petit échantillon de sang pour évaluer plusieurs paramètres épigénétiques. Elle s’est appuyée sur une méthode d’imagerie de molécules individuelles qu’elle avait mise au point lors de ses recherches postdoctorales à la Harvard Medical School et au Broad Institute. Cette méthode permet de réaliser une cartographie épigénétique précise avec seulement une très petite quantité de matière première, à l’aide d’un microscope fluorescent. Elle peut être utilisée, par exemple, pour visualiser les marques épigénétiques sur les nucléosomes, des morceaux d’ADN enroulés autour de « bobines » de protéines. Ces derniers peuvent être rejetés dans la circulation sanguine comme des morceaux d’épaves lorsque les cellules sont détruites. Le docteur Shema a donc pensé que les millions de nucléosomes présents dans le sang pourraient être analysés pour détecter le cancer.
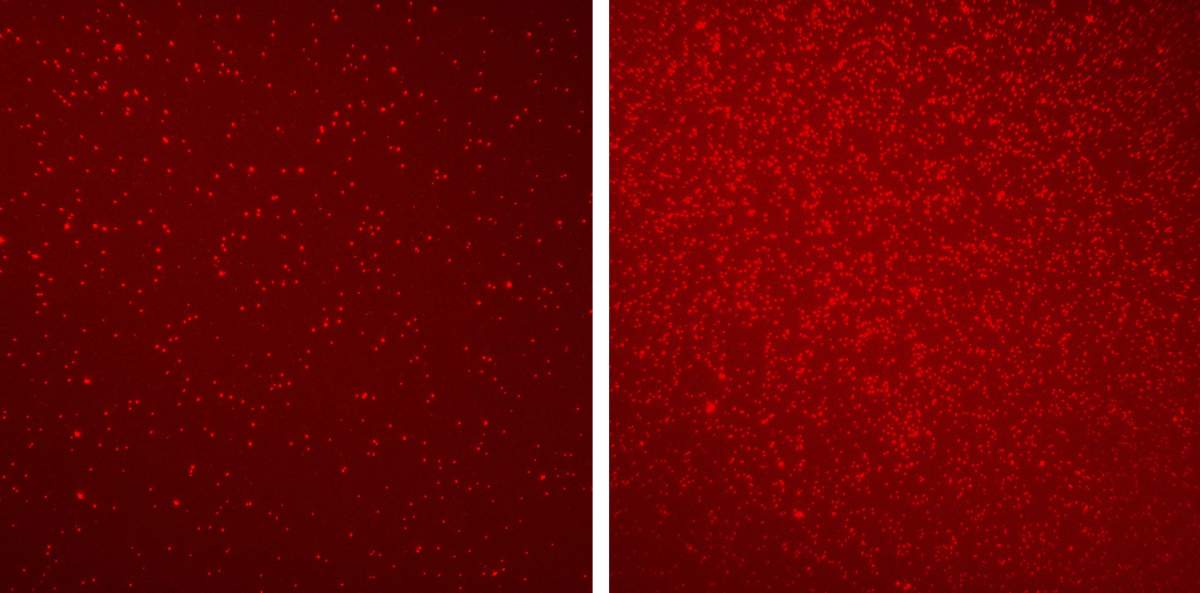
Différents profils de marqueurs épigénétiques révélés par EPINUC sur les nucléosomes sanguins (points rouge vif) d’une personne en bonne santé (à gauche) et d’un patient atteint de cancer colorectal (à droite).
À l’aide de la méthode d’imagerie monomoléculaire du docteur Shema, V.Fedyuk et N.Erez, ainsi que leurs collègues, ont comparé les nucléosomes dans le sang de 30 personnes en bonne santé à ceux de 60 patients atteints de cancer colorectal à différents stades. Ils ont constaté que les nucléosomes des deux groupes étaient caractérisés par des schémas de marquage épigénétique très différents. Cette analyse a porté sur six modifications épigénétiques différentes liées au cancer, ainsi que sur une variété d’autres indicateurs de cancer, notamment des segments de protéines provenant de tumeurs mortes, qui sont indétectables par les technologies conventionnelles.
Ensuite, en collaboration avec le professeur Guy Ron de l’Institut de physique Racah de l’Université hébraïque de Jérusalem, les scientifiques ont combiné ce qu’ils avaient révélé sur la biologie moléculaire du cancer avec des algorithmes d’intelligence artificielle, en appliquant l’apprentissage automatique aux grands ensembles de données obtenus des deux groupes. L’analyse a été effectuée non seulement sur tous les marqueurs du cancer, mais aussi sur les combinaisons et les relations entre eux. Pour s’assurer que leurs résultats ne se limitent pas au cancer colorectal, les scientifiques ont également appliqué leur technologie pour comparer les nucléosomes sanguins de volontaires sains à ceux de dix patients atteints d’un cancer du pancréas.
« Notre algorithme a pu faire la différence entre le groupe de patients sains et le groupe de patients avec un niveau de certitude record pour des études de ce type – avec une précision de 92 % », déclare le docteur Shema. Les scientifiques appellent cette nouvelle technologie EPINUC, un acronyme pour « épigénétique des nucléosomes isolés du plasma ».
S’ils sont confirmés par des études portant sur un plus grand nombre de patients, ces résultats pourraient déboucher sur un test sanguin multiparamétrique permettant de détecter et de diagnostiquer un cancer en utilisant moins d’un ml de sang. À l’avenir, en raison du niveau de détail révélé par l’analyse, les résultats de ce test sanguin pourraient également faire progresser la médecine personnalisée en suggérant les meilleurs traitements pour chaque patient.
Le docteur Shema résume : « Nous avons obtenu une preuve de concept réussie pour notre méthode, qui doit maintenant être confirmée par des essais cliniques. À l’avenir, notre approche multiparamétrique pourrait servir à diagnostiquer non seulement divers cancers mais aussi d’autres maladies qui laissent des traces dans le sang, comme les troubles auto-immuns ou les maladies cardiaques. »
La Science en chiffres
Chaque cellule de notre corps contient de l’ADN composé d’environ 6 000 000 000 de bases, qui sont empaquetées dans quelque 30 000 000 de nucléosomes. Chaque nucléosome porte de multiples modifications épigénétiques qui peuvent se présenter sous plus de 100 combinaisons différentes.
Les recherches du Dr Efrat Shema sont soutenues par l’Institut de la Société Suisse pour la Recherche sur la Prévention du Cancer et l’Institut Henry Chanoch Krenter pour l’Imagerie Biomédicale et la Génomique. Le Dr Shema est titulaire de la Chaire de Développement de Carrière de la Famille Lisa et Jeff Aronin.


